|
O sequenciamento de DNA é um processo que determina a
ordem dos nucleotídeos (blocos que constituem a molécula de DNA) em uma
amostra. Existem vários métodos disponíveis, e cada um apresenta vantagens
e desvantagens.
Um dos mais utilizado é o chamado “método didesoxi”
conhecido também como de “terminadores de cadeia” ou de “Sanger”; ele
constitui a base da metodologia empregada no sequenciamento do genoma
humano. Sua estratégia consiste em identificar, continuamente e
sequencialmente durante o processo, o último nucleotídeo
incorporado na extremidade de alongamento da cadeia. Os produtos da reação
deverão também portar uma “marca” que permita detecta-los na etapa de
análise. Resumidamente, o processo é realizado a partir de uma cadeia
simples (não dupla) do DNA a ser sequenciado; esta servirá de molde para
gerar a outra metade complementar da dupla hélice. Isto é obtido pela
desnaturação da “molécula nativa” (fig 1).
FIG 1

A reação de síntese se processa em condições iônicas e de
pH apropriadas, na presença da enzima DNA polimerase e de uma mistura dos
4 nucleotídeos sob a forma de 3’-desoxinucleotídeo trifosfatos (dNTPs):
dATP, dCTP, dGTP e dTTP sendo um deles marcado radioativamente com ³²P ou
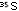 ; um dos
mais utilizado é o dATP ; um dos
mais utilizado é o dATP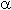 ³²P (fig 2a). Como a
enzima utilisada catalisa somente o alongamento da cadeia nascente é
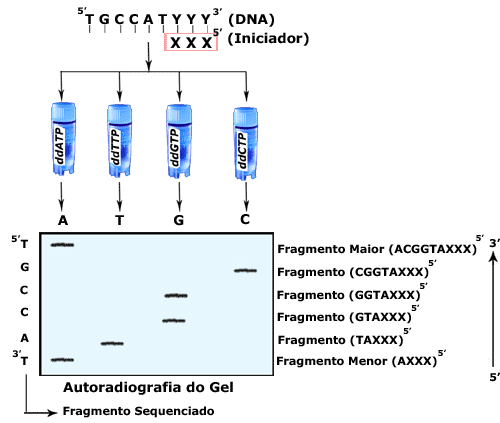
necessário também, a presença na reação, de um pequeno fragmento de DNA
sintético (XXX) complementar a uma região conhecida (YYY)
na extremidade 3’ do DNA molde; estas características permitirão sua
hibridização no local mencionado, e fornecerão um ponto de partida para a
replicação do DNA. O fragmento XXX, denominado iniciador ou “primer",
quando marcado, poderá ser utilizado para rastrear o fragmento de DNA
recém sintetizado no lugar do dNTP. Fato importante no processo é que ele
é executado em quatro reações separadas; cada uma delas,
contendo adicionalmente pequena quantidade de um (e
apenas um) dos 4 tipos de cada dNTP sob a forma de análogo, 2’,
3’-didesoxinucleotídeo trifosfatos (ddNTPs) (fig 2b). ³²P (fig 2a). Como a
enzima utilisada catalisa somente o alongamento da cadeia nascente é
necessário também, a presença na reação, de um pequeno fragmento de DNA
sintético (XXX) complementar a uma região conhecida (YYY)
na extremidade 3’ do DNA molde; estas características permitirão sua
hibridização no local mencionado, e fornecerão um ponto de partida para a
replicação do DNA. O fragmento XXX, denominado iniciador ou “primer",
quando marcado, poderá ser utilizado para rastrear o fragmento de DNA
recém sintetizado no lugar do dNTP. Fato importante no processo é que ele
é executado em quatro reações separadas; cada uma delas,
contendo adicionalmente pequena quantidade de um (e
apenas um) dos 4 tipos de cada dNTP sob a forma de análogo, 2’,
3’-didesoxinucleotídeo trifosfatos (ddNTPs) (fig 2b).
FIG 2
Estes análogos conhecidos como “terminadores” quando
incorporados à cadeia nascente, por não apresentarem 3’OH que permita
formar ligação com o próximo dNTP a ser adicionado, bloqueará todo
processo. Como todos os nucleotídeos normais (dNTPs) estão presentes, o
alongamento da cadeia prosseguirá até que a enzima DNA-polimerase insira
um análogo (ddNTP). Consequentemente, haverá parada imediata da reação de
síntese no ponto em que seu alongamento foi interrompido: na extremidade
e, a molécula estará marcada com ³²P. Os fragmentos assim obtidos, cada
qual contendo um resíduo final conhecido, pois se sabe em que tubo de
reação o análogo foi adicionado, são separados por tamanho em gel de
poliacrilamida individualmente: um canal de análise para cada reação. O
gel é “transferido” para um suporte de nitrocelulose (filtro).
Após
auto-radiografia a ordem dos nucleotídeos, na cadeia de DNA recém
sintetizada, pode ser visualizada e obtida diretamente; esta é
complementar à da molécula sequenciada: que serviu de
“molde”. Levando estas observações em consideração, é possível conhecer a
sequência de nucleotídeos do DNA sequenciado de 3’para 5’ partindo-se da
parte inferior para a superior do gel. O resultado e as etapas do processo
descrito acima, frequentemente mostrado em fotografias nos livros
especializados, podem ser observados no esquema da Fig. 3.
FIG 3
 |
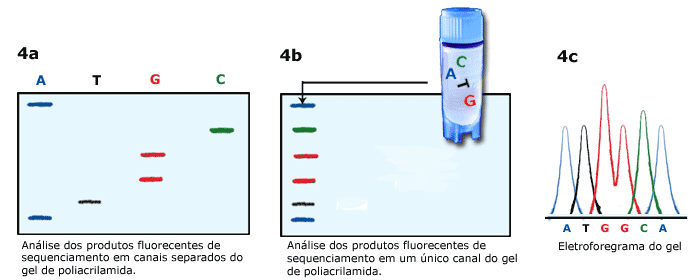
O método pode ser automatizado através de
“maquinaria apropriada” gerenciada por computadores com “softs” que
lêm sequencialmente e identificam os produtos. Isto permitirá
executar o processo em grande escala. Neste caso utilizam-se
simultaneamente os 4 didesoxinucleotídeos terminadores (ddNTPs)
marcados por fluorescência fig 4a. Como cada reação (A,T,G,C)
utilizou um fluorocromo diferente os produtos podem ser reunidos e a
eletroforese destes realizada em um único canal do gel de
sequenciamento fig 4b. O sinal fluorescente diferencial emitido por
cada fragmento, após iluminação com um feixe de laser, identificará
os produtos baseado na diferença de comprimento de onda. A luz
emitida é detectada por “escaneamento” do gel e a sequência deduzida
por computador fig 4c. Variáveis mais modernas, consequentemente
mais rápidas e poderosas, incluem a robotização total do processo
com a inclusão das etapas de purificação e da reação de síntese da
cadeia do DNA |
FIG 4
 |

